L’ADN mitochondrial est localisé dans les mitochondries. C’est un ADN circulaire de 16569 nucleotides. Hommes et femmes ont de l’ADN mitochondrial ; tout homme a le même ADN mitochondrial que sa mère, par contre, il ne transmet pas cet ADN mitochondrial à ses enfants (qui auront celui de leur mère). En effet, il n’y a pas de mitochondries dans les spermatozoides ; les mitochondries sont uniquement dans l’ovocyte. Les lignées d’ADN mitochondrial sont des lignées de mère en fille. Comme pour le chromosome Y les mutations permettent de définir des haplogroupes. Nous allons étudier l’haplogroupe le plus fréquent en Europe, l’haplogroupe H , ce qui sera un bon support pour montrer les méthodes
et les résultats.
Carte de l’haplogroupe H :
Cette carte appelle certains commentaires. On présente ici les résultats combinés de tous les sous-groupes connus du grand haplogroupe H. Sur la carte ci-dessus on voit un groupe fort en Europe qui semble diffuser sur l’Asie et l’Afrique du nord. Voici ma grille de lecture de cette carte : l’haplogroupe H est absent des Amériques, absent d’extrême orient (chine, japon) et d’Océanie, et l’haplogroupe H est absent de l’Afrique noire. Je trouve remarquable que les populations sont de l’haplogroupe H presque en proportion de leur proximité au type blanc européen. Il n’y a pas d’explication mais je trouve que ce constat suffit à souligner l’intérêt de ces analyses.
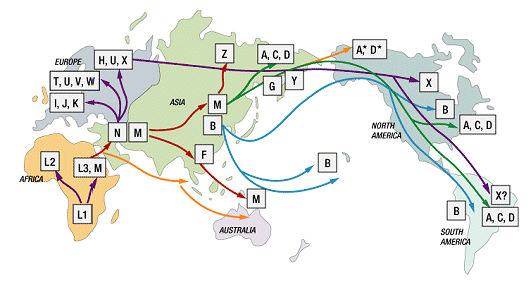
Situons maintenant l’haplogroupe H dans l’arbre schématique ci-dessous. H est l’une des branche du groupe R0, dans la grande division N. Sur cet arbre les groupes « ancestraux » L0, L1, L5, L2, L6 et L4 sont tous uniquement africains. On constate que seuls des descendants de L3 sont à l’origine de tous les haplogroupes du reste du monde (hors Afrique). C’est cette constatation qui est a l’origine de la théorie « Out of Africa » : les groupes humains se sont développés dans un premier temps en Afrique et, à partir de L3, il y a sortie d’Afrique. En fait on constate ce qui peut s’interpréter comme une double sortie d’Afrique : branches M et N. La branche M correspondrait à une « route sud », sans doute par cabotage, le long des côtes d’Arabie puis de l’Inde, vers l’Asie du sud Est, l’Océanie. La branche N aurait d’abord investi la péninsule arabique qui jouissait d’un climat plus humide, puis, de ce creuset il y aurait eu expansion vers le moyen orient, l’Europe et l’Asie.
Les haplogroupes fréquents en Europe sont H, V, J, T K, U, plus rarement I,W, et X. Ce sont là de grandes divisions mais un haplogroupe comme H comporte plus de 10 branches d'importances inégales. Quelques cartes de sous-groupes :
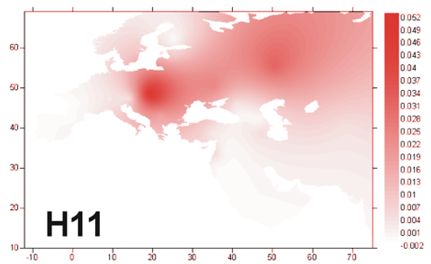
J'ai choisi 2 sous groupes dont la dispersion est presque opposée : H1 est plus faible là où H11 est fort. H1 est, de loin, le sous groupe le plus important d'Europe de l'ouest. Voici 2 autres cartes :
Attention, les échelles de coloration ne sont pas les mêmes d'une carte à l'autre. Le groupe H7 est bien moins fréquent que le groupe H1 en France (environ 5 fois moins).
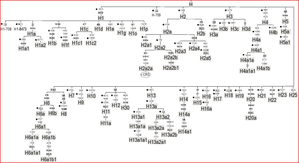
Arbre des sous-groupes mitochondriaux H :
Sur cet arbre sont indiqués les mutations qui définissent les sous-groupes par leur position, entre 1 et 16569, sur l'ADN mitochondrial . Cet arbre montre la variété de l'ADN mitochondrial, rien que pour l'haplogroupe H. Ainsi, la première carte pour l'ensemble de l'haplogroupe H recouvre une réalité variée. Voici une autre carte qui permet de bien voir que la prédominance de l'haplogroupe H1 est spécifique à l'Europe :

L'haplogroupe H1 (en rouge) fait place à un « cocktail » de sous-groupes au moyen orient. On interprète ceci comme une origine probable de H au moyen orient, peut être au Caucase. Seuls certains sous-groupes, dont H1, sont « partis à la conquête de l'Europe ».
On peut reprendre les données précédentes dans un arbre plus général, re-situant l'haplogroupe H dans l'ensemble des groupes mitochondriaux avec des mutations qui structurent cet arbre :
Arbre mitochondrial :
On a établi qu’une mutation se produit environ tous les 4000 à 5000 ans. C’est très approximatif mais ça permet de tirer un certain nombre d’informations. Les souches N et M telles que nous les connaissons aujourd’hui sont le résultat d’une longue période d’évolution ; les mutations de la souche M pouvant correspondre à 16000 – 20000 ans et les mutations de la souche N à 20000 – 25000 ans. Il y a donc eu une lente maturation.
La mention « rCRS » qui signifie « revised Cambridge Reference Sequence » se trouve être un ADN mitochondrial du sous groupe H2a2a, qui a été choisi au début des études comme ADN de référence. Nous allons utiliser les mutations rapportées pour montrer comment on peut schématiquement évaluer l’âge d’un groupe, ou plus exactement d’un nœud de l’arbre, ici R . Entre rCRS et R on peut compter 11 mutations et sur la branche U il y aurait un nombre semblable de mutations jusqu’au sous-groupe U5b1b qui est un groupe Européen. L’ancêtre commun R vivait il y a 44000 – 55000 ans et avec les 20000 ans précédents on comprend comment la « sortie d’Afrique » est datée de 60000 – 70000 ans. Alors que U est un groupe qui semble avoir été européen dès le début, R0 est un groupe du moyen orient où HV, puis H et V se différencient ultérieurement.
Les mutations en bleu sur l'arbre ci-dessus sont les mutation de la région autour de l'origine de l'ADN mitochondrial : nucléotides 16024 – 16569 (=0) et 1 – 576. Sur cette section d'environ 1000 nucléotides (par rapport à 16569 pour la taille totale) le taux de mutation est plus important (mais la zone est relativement petite). On observe que sur une durée estimée de 10000 ans 3 mutations vont se produire : 1 dans la région de contrôle (la zone de 1000 nucléotides), et 2 dans la région dite « région codante » (tout le reste de l'ADN mitochondrial).
Les analyses d'ADN mitochondrial sont arrivées à l'âge adulte depuis que toute la molécule , les 16569 nucléotides, peuvent être séquencés et analysés de façon systématique. L'ensemble des ADNs ainsi entièrement séquencés dépasse maintenant 20000 ; on n'est plus limité dans les comparaisons par le manque de données.
La limite actuelle c'est l'interprétation quand on descend au niveau des sous-groupes. L' origine de H est daté d'environ 18000 ans mais de nombreux sous groupes semblent bien plus récents. Une caractéristique de l'ADN qu'il faut garder en tête est qu'il y a un « pool » important de personnes qui sont H ou dites H* c'est à dire avec 1 mutation (sans doute familiale) supplémentaire seulement. C'est ce qui est observé et le fait qu'il y ait des branches ayant accumulé de nombreuses mutations alors qu'un ensemble important de personne conserve l'ADN H d'origine est une difficulté d'interprétation. Selon les auteurs H1 sera Magdalénien (-14000) ou Indo-Européen (premiers néolithiques -9000). On attend la bonne méthode pour analyser ces données. Pendant ce temps les données augmentent et les cartes se précisent. Pour revenir à la carte qui a servi d'introduction on peut dire que l'origine de H est sans doute vers le Caucase mais qu'il y a eu sur la longue période considérée une conquête de l'Europe et sans doute diffusion à partir de l'Europe, peut être avec les fameux Indo-Européens, vers le moyen orient, jusqu'à l'Inde. La zone nord-africaine a une répartition en sous-groupes comparable à l'Europe (sud) ce qui souligne la perméabilité géographique dans ces temps anciens.